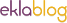-
Perl répétitions des bases: aléas.
27.02.16 Paris
# Comptage, par aléa, des répétitions de bases pour un %GC donné.
# 3 paramètres à changer ici sur la 1ère ligne et sauvegarder avant d'exécuter le programme dans un terminal.
# exemple compter les répétitions pour un %GC de 17/46=36.9% et un chromosome de 5 013 479 bases.
my $ll=46; $gc=17; $efc=5013479;
my $at=$ll-$gc;
my $gc1=$gc-1; $at1=$at-1;
my $gc2=$gc+3; $gc3=$gc2+1; $gc4=$gc3+$gc-2;
my $at2=$gc4+1; $at3=$at2+$at-2; $at4=$at3+1; $at5=$at4+$at-2;
my $l=$ll*2; # le max de l'aléa ***
my $i=0;
my $r=0;
my $k=0;
my $s=0;
my $v=0;
my $a=0;
my $e=0;
# Initialisation du tableau pour les fréquences
@Valeurs = ();
foreach $b (2..5) {foreach $n (2..21) { $Valeurs [$b] [$n] = 0;}} # ********* augmenter les bornes supérieures si nécessaire: séquences spécifiques ************
my @m= (1,2,3,4);
my @p = (0)x4;
#
my @u=(1,$at2..$at3); @d=(2,$at4..$at5); @t=(3,5..$gc2); @q=(4,$gc3..$gc4);
for ($k=1; $k<=$efc; $k+=1) {
$r = int rand($l) + 1;
#print ($r, "\n");
if ($s==0) {
foreach $i (0..$at1) { if ($r == $u[$i]) {
$s=1;
}}}
if ($s==0) {
foreach $i (0..$at1) { if ($r == $d[$i]) {
$s=2;
}}}
if ($s==0) {
foreach $i (0..$gc1) { if ($r == $t[$i]) {
$s=3;
}}}
if ($s==0) {
foreach $i (0..$gc1) { if ($r == $q[$i]) {
$s=4;
}}}
Freq($s);
if($v == 0 ) {
if($a==0) {$a=$s;}
if($s==$a) { $e +=1;} else {$v=1; }}
$s=0;}
$s=0;
Freq($s);
#
sub Freq {
#
my ($s) = @_;
my $j=0;
# ********* Comptage de toutes les séquences répétitives. A compléter pour les séquences spécifiques. ************
#
if( $s == 1 ) {
if($p[1] == 0) {
foreach $j (@m, $m[$j]!=1) {
if ( $p[$j] !=0 ) { $n=$p[$j]+1; $b=$j+1;
$Valeurs [$b] [$n] +=1; $p[$j]=0; }}
$p[1]+=1;}
else { $p[1]+=1;}}
if( $s == 2 ) {
if($p[2] == 0) {
foreach $j (@m, $m[$j]!=2) {
if( $p[$j] !=0 ) { $n=$p[$j]+1; $b=$j+1;
$Valeurs [$b] [$n] +=1; $p[$j]=0; }}
$p[2]+=1;}
else {$p[2]+=1;}}
if( $s == 3 ) {
if($p[3] == 0) {
foreach $j (@m, $m[$j]!=3) {
if( $p[$j] !=0 ) { $n=$p[$j]+1; $b=$j+1;
$Valeurs [$b] [$n] +=1; $p[$j]=0; }}
$p[3]+=1;}
else {$p[3]+=1;}}
if($s == 4 ) {
if($p[4] == 0) {
foreach $j (@m, $m[$j]!=4) {
if( $p[$j] !=0 ) { $n=$p[$j]+1; $b=$j+1;
$Valeurs [$b] [$n] +=1; $p[$j]=0; }}
$p[4]+=1;}
else {$p[4]+=1;}}
if($s != 0 ) { return}
if($s == 0 ) {
foreach $j (@m) {
if ( $p[$j] !=0 ) { $n=$p[$j]+1; $b=$j+1;
$Valeurs [$b] [$n] +=1; $p[$j]=0; } }}
@c=(1..4);
foreach $k (0..3) {
if ($c[$k] == $a) { $x=$k+2;}};
if ( $x == $b ) {
$Valeurs [$b] [$n] -=1;
$n = $n + $e;
$Valeurs [$b] [$n] +=1;
$n = $e+1;
$Valeurs [$b] [$n] -=1;}
#
#print ("e=", $e, "\n");
print ("n t a c g \n");
foreach $k (2..21) { $j=$k-1; print ( $j," ", $Valeurs [2] [$k]," ", $Valeurs [3] [$k]," ", $Valeurs [4] [$k]," ", $Valeurs [5] [$k], "\n")};
return;}
#« Les états vibratoires de l'ADN et la sélection naturelle: tableauxPerl répétition des bases: chromosome »
-
Commentaires